Integrando conhecimentos em uma escola técnica federal: aprendendo a utilizar ferramentas tecnológicas e conceitos matemáticos nos mistérios da expressão de genes
Luciana Ferrari Espindola Cabral
Mestre em Botânica, docente (Cefet-RJ, Uned Maria da Graça)
Alan Nunes de Albuquerque
Estudante do curso técnico em Segurança do Trabalho (Cefet-RJ, Uned Maria da Graça)
Thays Meirelles Borges de Azevedo
Estudante do curso técnico em Segurança do Trabalho (Cefet-RJ, Uned Maria da Graça)
Larissa Brillo Nunes Rubio
Estudante do curso técnico em Segurança do Trabalho (Cefet-RJ, Uned Maria da Graça)
Fabiana Cordeiro
Doutora em Microbiologia, docente, coordenadora do Ensino Médio Integrado (Cefet-RJ, Uned Maria da Graça)
Introdução
O DNA ou ADN (ácido desoxirribonucleico) carrega nossas informações genéticas e em sua estrutura são encontrados os nucleotídeos, que são compostos por bases nitrogenadas (adenina, timina, citosina e guanina), além de um açúcar (a desoxirribose) e um grupamento fosfato. Os genes são unidades compostas por DNA, possuindo a função de transmissão das características hereditárias; é importante ressaltar que cada um deles codifica uma determinada cadeia polipeptídica ou RNA funcional e é responsável pela geração de uma característica ou pela realização de determinada função dentro da célula.
Sabendo disso, o que é a expressão gênica e qual a relação deste artigo com esse conceito? Primeiro: a expressão gênica consiste na produção de RNAm (RNA mensageiro), conhecida como transcrição, e a posterior síntese da proteína chamada tradução.
Vandesompele et al. (2002) afirmam que
analisar a expressão gênica é importante em muitos campos da pesquisa em Biologia. Entender padrões dos genes expressos é importante para elucidar mecanismos regulatórios e levar a identificação de genes relevantes a novos processos biológicos ou implicados em doenças.
Em 1993, Kary Mullis ganhou o Prêmio Nobel de Química por desenvolver a técnica conhecida como Reação em Cadeia da Polimerase (PCR), em que é utilizado um equipamento capaz de amplificar in vitro sequências de DNA curtas, utilizando variações de temperatura e fragmentos de DNA sintetizados em laboratório, chamados iniciadores ou primers (em inglês).
Os pesquisadores trabalham com a ferramenta denominada Reação em Cadeia da Polimerase de Tempo Real (RT-PCR), que consiste em amplificar o DNA cópia de uma sequência específica e quantificar a expressão do produto durante o progresso da PCR, utilizando um termociclador capaz de medir a fluorescência emitida quando o DNA é amplificado. O DNA cópia é obtido utilizando uma enzima especial, a transcriptase reversa, que converte o RNAm em cDNA (DNA cópia). A quantificação acontece por meio de fluorescência, dentro do sistema que utiliza sondas (que emitem fluorescência) e bloqueadores de fluorescência (chamados de quencher) ou intercalante (molécula que se liga ao DNA de fita dupla, ou seja, amplificado).
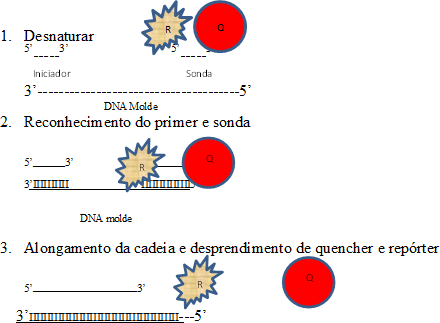
Figura 1: Mecanismos de anelamento de primers e sondas em uma reação de PCR em tempo real. O R da figura é a molécula que emite a fluorescência e está ligado fisicamente com o quencher em 1. Nessa configuração, o quencher bloqueia a emissão da fluorescência. Em 2, quando a sonda e o primer se combinam com a cadeia de DNA, inicia-se um processo de liberação de primer e sonda, que pode ser visualizado em 3. Uma vez livre o R (chamado de repórter), pode emitir a fluorescência que é detectada pelo termociclador real-time
Fonte: Fabiana Cordeiro (2017).
Existem dois métodos de quantificação: a absoluta e a relativa. Na absoluta, o pesquisador é capaz de determinar exatamente, por exemplo, quantas cópias de um vírus existem na amostra. Na quantificação relativa, determinamos mudanças na expressão de um gene em uma dada amostra em relação a uma amostra de referência. Este método inclui Ct (ΔΔCt) comparativo e quantificação relativa, em que determinamos quanto daquele gene é expresso em relação a outro tem em cada amostra. Esse método utiliza uma amostra calibradora para comparação (Veja em: http://www.dorak.info/genetics/realtime.html). O Ct (Ciclo Threshold) reflete o número do ciclo no qual a fluorescência gerada em uma reação cruza o threshold. Na prática, reflete o número de amplicons que foram acumulados.
A estratégia de cálculo que será utilizada neste caso para quantificação relativa é o método CT comparativo, em que se aplica a fórmula aritmética 2-ΔΔCT com o fim de comparar mudanças na expressão de genes.
Anteriormente a ela, deve-se estabelecer uma relação entre o gene alvo a ser analisado e o controle endógeno 1 que serve como referência ativa para normalizar os valores de quantificação; este é um gene utilizado para normalizar a quantificação de um RNA mensageiro alvo para corrigir a quantidade de RNA adicionada à reação. Após a subtração do CT alvo 2 com o CT endógeno 3 obtém-se o ΔCT que servirá para ser comparado com o ΔCT calibrador 4 referente à amostra calibradora escolhida, utilizada para comparar a expressão gênica entre amostras. Dessa comparação será obtido o ΔΔCT que será o recurso utilizado para diferenciar a expressão gênica nas situações em estudo.
Logo, o propósito deste material é entender o processo de expressão gênica de forma didática e como ele pode ser utilizado laboratorialmente para detectar a expressão de determinado gene. Para facilitar esse processo, propomos a utilização de um estudo de caso hipotético de três surtos causado por bactéria E.coli O157:H7, que produz a toxina Stx, que se liga a receptores das células renais, paralisando e causando a necrose dos rins. Para esse fim, o aluno será o pesquisador-investigador que receberá três hambúrgueres de redes de fast food de três cidades diferentes; em cada uma, a toxina agiu de uma forma, causando mais ou menos mortes.
Desenvolvimento
O aluno será o pesquisador e receberá informações sobre hambúrgueres de três cidades diferentes: Rio de Janeiro, Porto Seguro, e Sakai, no Japão. Em Porto Seguro, foi informado ao aluno/aluna que foram registradas mortes relacionadas à doença com paralisia da função renal.
Após confirmar que a bactéria envolvida é a E.coli O157:H7, o pesquisador resolveu estudar a presença da toxina Stx2a, que paralisa a função renal. Ele deverá utilizar diversos bancos de dados; o principal utilizado para obtenção de resultados e realização de simulações é o site do NCBI (National Center for Biotechnology Information).
Para realizar a PCR em tempo real, já que o pesquisador quer ver a expressão diferencial da toxina nos três surtos, é necessário que tenha determinado diversos parâmetros (descritos mais à frente) e tenha posse do material necessário. Utilizando o site NCBI, ele deve determinar quais serão os primers e sondas do gene alvo e do controle endógeno.
Primer, também conhecido como iniciador, é o dispositivo utilizado na PCR para sintetizar a sequência alvo selecionada; para isso há o forward primer (5' - 3'), que segue o sentido normal de síntese da cadeia (Figura 1), e o reverse primer, seguindo o sentido inverso. A região flanqueada pelos dois primers (o forward e o reverse) é que vai determinar a sequência de DNA a ser amplificada.
Atividade 1: Achando sequências no NCBI
O procedimento para determinação de primers e sondas para a toxina que se quer pesquisar segue a seguinte ordem:
1) Acessar o site NCBI, no link https://www.ncbi.nlm.nih.gov/; ao lado do campo de pesquisa, deve-se selecionar o objeto específico de procura; neste caso, gene, após digitar o nome da toxina "stx2A" no campo de pesquisa.
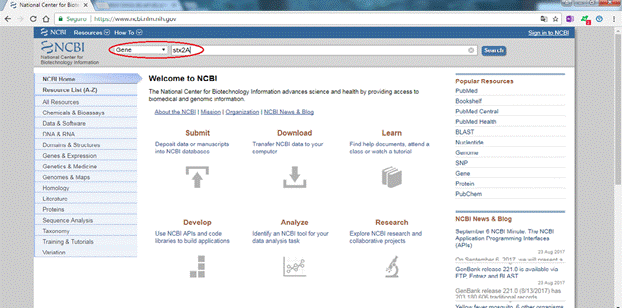
Figura 2: Passo a passo de obtenção de sequências no site NCBI
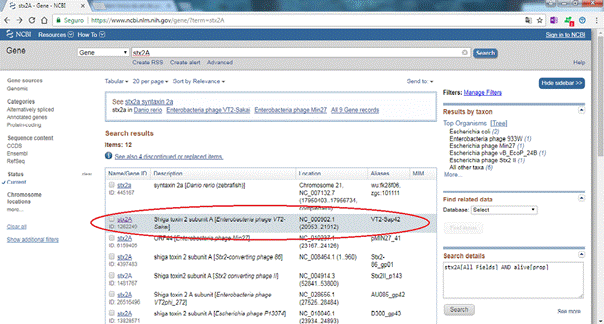
Figura 3: Achando a sequência específica da toxina
2) Procurar o código ID: 1262249 (referente a E.coli O157:H7) e selecionar o gene referente a ele; diversos dados serão mostrados, mas para determinar os primers deve-se selecionar a opção FASTA, em que é mostrada a sequência específica do gene stx2a, ao lado selecionar a opção "pick primers".
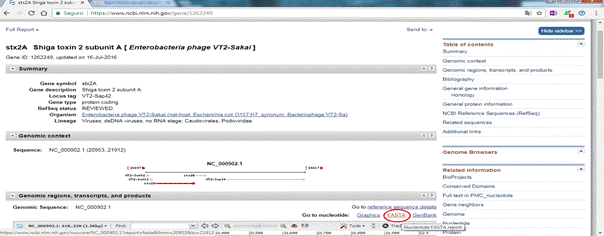
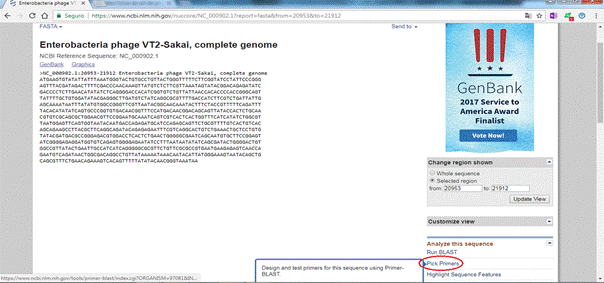
Figuras 4 e 5: O formato FASTA mostra exatamente a sequência de bases nitrogenadas que formam o DNA.
3) Uma caixa para preenchimento de dados é mostrada; é necessário modificar apenas o valor máximo da caixa "PCR product size" para até 150pb o amplicon 5 e mudar a pesquisa da caixa "Database" para "nr" em seguida selecionar "Get primers"; então selecionar a caixa ao lado da sequência AP000422.1 e clicar em Submit no final da página. O primer a ser selecionado é o mostrado em "Primer pair 1". O pesquisador utilizará as sequências apresentadas pelo programa: GGACCACATCGGTGTCTGTT (forward primer) e ATTAACGAACCCGGCCACAT (reverse primer), que corresponderão ao primer/iniciador específico da toxina.
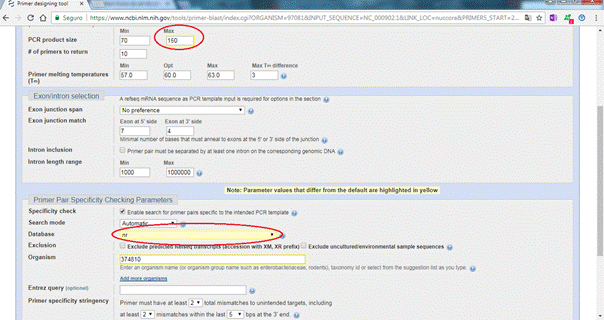
Figura 6: Como desenhar primers (forward e reverse) utilizando a base de dados NCBI, respeitando os critérios dados pelo real time.
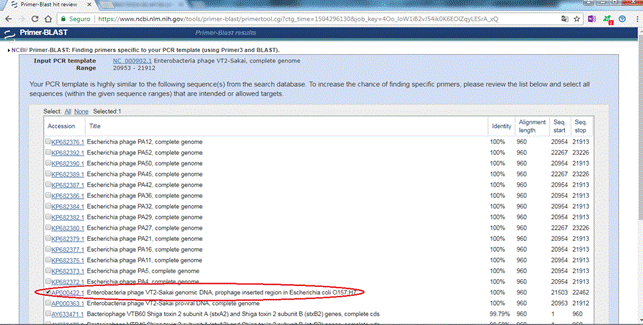
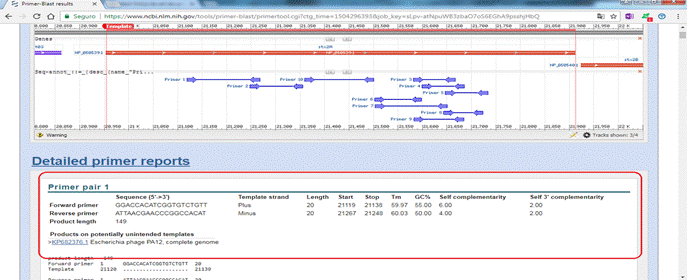
Figuras 7 e 8: Passo a passo para obtenção de primers/iniciadores para o real time PCR.
O pesquisador deve repetir o procedimento acima para o controle endógeno pesquisando por "O157:H7 16S", apenas levando em consideração que o código do gene a ser selecionado nesse caso é ID: 914974, sendo escolhido o gene rrsB (16s; controle endógeno). O primer a ser selecionado segue o esquema citado; as sequências apresentadas pelo programa são GGCGCATACAAAGAGAAGCG (forward primer) e CTCCAATCCGGACTACGACG (reverse primer).
Para determinação das sondas de ambos (gene alvo e controle endógeno), o processo a ser seguido envolve algumas regras: uma sonda deve estar situada próximo ao primer forward (distância de geralmente 1 a 5 bases); em geral, o conteúdo de guaninas e citosinas da sonda deve estar entre 35 e 65% e não se deve ter uma guanina no final 5' (próximo ao fluorocromo reporter) da sonda (Dorak, 2006). Todo esse processo será realizado manualmente, ou seja, o pesquisador deverá, a partir das sequências dos genes stx2a e 16S, localizar os primers determinados anteriormente e determinar as sondas, sendo elas: TATCAGTGCC para o gene alvo e CTGCCAGTGA para o controle endógeno (16S).
Determinados os primers e sondas, o próximo processo a ser feito é a conversão de RNA em cDNA. Para que esse processo ocorra, é necessário que haja primeiramente a extração do RNA alvo; no caso deste experimento, os RNAs dos três hambúrgueres relativos a cada um dos surtos hipotéticos. Após a extração, o RNA é tratado com uma DNase I para eliminação de qualquer resquício de DNA genômico, é dosado e verificada sua qualidade e integridade. Além disso, é preciso dosar esse RNA, porque a mesma quantidade do material deve ser colocada entre as amostras para evitar variação de quantidade entre os materiais, o que levaria a erros de interpretação. Em seguida, como indicado na Figura 9, acontece o processo de conversão do RNA em cDNA, com posterior determinação da expressão do gene em um único tubo/etapa.
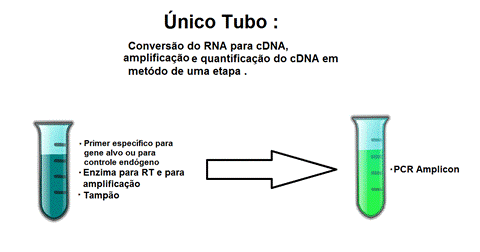
Figura 9: Inserção de RNA quantificado em um tubo, primer específico para gene alvo ou endógeno, enzima RT para converter RNA em DNA cópia; tampão; nucleotídeos e sondas específicas para cada iniciador. Em seguida, desnaturar (abrir) as fitas, parear os iniciadores/primers (caso haja correspondência) e extensão da cadeia
Fonte: Thays Azevedo (2017).
Atividade 2: Desvendando a expressão gênica
Trabalhando com os conceitos vistos, a amostra calibradora, que será utilizada para intuito de comparação, será a amostra 1 (do Rio de Janeiro). O aluno receberá do equipamento de real time, os seguintes resultados em triplicata (média + desvio padrão) de cada Ct:
Tabela 1: Resultados obtidos pelo PCR em tempo real (ou real-time PCR) para análise da expressão gênica
| Stx2a | 16s | ΔCt | ΔΔCt | 2-ΔΔCt | |
| Surto 1 | 30,99 + 0,15 | 23, 69 + 0,05 | 7,3+0,17 | 0,00 + 0,17 | 1,0+ 0,11 |
| Surto 2 | 25,83 + 0,07 | 23,01 + 0,07 | 2,82+ 0,10 | -4,48 + 0,10 | 22,31 +1,10 |
| Surto 3 | 27,03 + 0,06 | 22,66 + 0,08 | 4,37+ 0,0 | -2,93 + 0,10 | 7,62 + 0,32 |
1) Calcular o ΔCt de cada um dos surtos, utilizando como parâmetro o fato de que o valor do Ct da toxina deve ser normalizado com o valor do Ct do 16S.
Resposta: na tabela.
2) Calcular o ΔCt (Ct-alvo-Ct-endógeno). Em seguida, calcular o ΔΔCt (ΔCt alvo normalizado - ΔCt calibrador). A fórmula 2-ΔΔCt auxilia na conversão do resultado em unidades relativas de expressão.
Resposta: na tabela.
3) Responder com base nos resultados obtidos: por que o surto 2 causou mais mortes que os outros?
Resposta: Como a toxina foi expressa em muito maior quantidade no surto 2, provavelmente causou muitos danos ao hospedeiro, paralisando de forma muito eficiente a função renal.
Referências
DORAK, M.T. Real-Time PCR (Advanced Methods Series). Oxford: Taylor & Francis, 2006. Disponível em: http://www.dorak.info/genetics/realtime.html. Acesso em: set. 2017.
VANDESOMPELE, J. O.; DE PRETER, K.; PATTYN, F.; POPPE, B.; VAN ROY, N.; DE PAEPE, A.; SPELEMAN, F. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome biology, 18; 3(7), Research 0034, Epub jun. 18, 2002.
1Controle endógeno: referência ativa que corrige pequenas diferenças de RNAm entre as amostras. Não deve variar em diferentes condições experimentais.
2Gene que se quer pesquisar.
3Referência ativa.
4Utilizada como base de comparação para verificar aumentos ou decréscimos de expressão. Como exemplo podemos comparar a expressão de determinado gene no cérebro (utilizando este como calibrador) e a expressão do mesmo gene no fígado.
5Amplicon: produto da amplificação de um gene dentro da sequência de primers (ou iniciadores) escolhida. Para real-time, é recomendável que esse amplicon tenha até 150 pares de bases.
Publicado em 09 de janeiro de 2018
Novidades por e-mail
Para receber nossas atualizações semanais, basta você se inscrever em nosso mailing
Este artigo ainda não recebeu nenhum comentário
Deixe seu comentárioEste artigo e os seus comentários não refletem necessariamente a opinião da revista Educação Pública ou da Fundação Cecierj.

